Partners use AlphaFold, the AI system recognised last year as a solution to the protein structure prediction problem, to release more than 350,000 protein structure predictions including the entire human proteome to the scientific community.
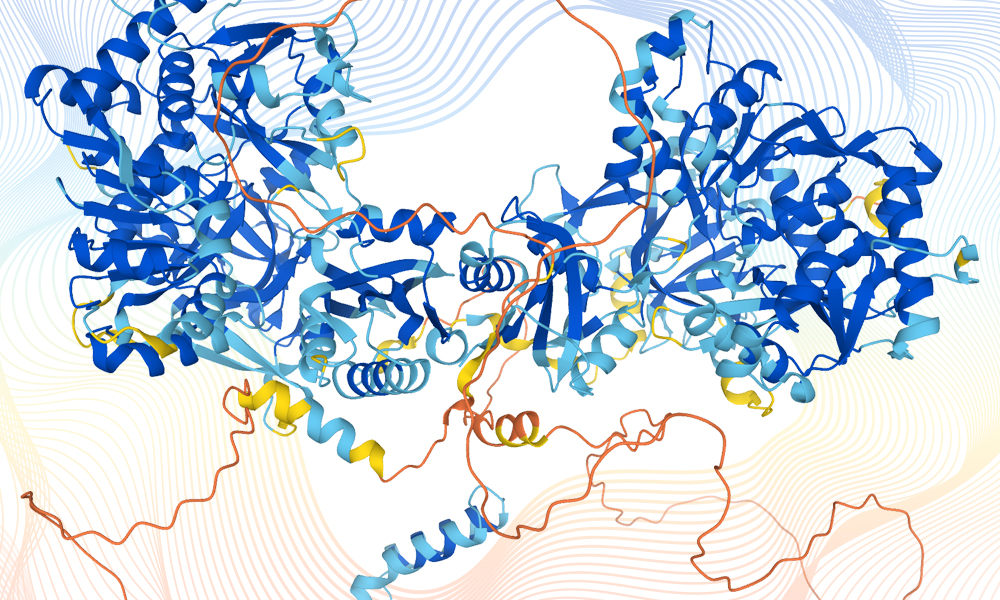
DeepMind today announced its partnership with the European Molecular Biology Laboratory (EMBL), Europe’s flagship laboratory for the life sciences, to make the most complete and accurate database yet of predicted protein structure models for the human proteome. This will cover all ~20,000 proteins expressed by the human genome, and the data will be freely and openly available to the scientific community. The database and artificial intelligence system provide structural biologists with powerful new tools for examining a protein’s three-dimensional structure, and offer a treasure trove of data that could unlock future advances and herald a new era for AI-enabled biology.
AlphaFold’s recognition in December 2020 by the organisers of the Critical Assessment of protein Structure Prediction (CASP) benchmark as a solution to the 50-year-old grand challenge of protein structure prediction was a stunning breakthrough for the field. The AlphaFold Protein Structure Database builds on this innovation and the discoveries of generations of scientists, from the early pioneers of protein imaging and crystallography, to the thousands of prediction specialists and structural biologists who’ve spent years experimenting with proteins since. The database dramatically expands the accumulated knowledge of protein structures, more than doubling the number of high-accuracy human protein structures available to researchers. Advancing the understanding of these building blocks of life, which underpin every biological process in every living thing, will help enable researchers across a huge variety of fields to accelerate their work.
Last week, the methodology behind the latest highly innovative version of AlphaFold, the sophisticated AI system announced last December that powers these structure predictions, and its open source code were published in Nature. Today’s announcement coincides with a second Nature paper that provides the fullest picture of proteins that make up the human proteome, and the release of 20 additional organisms that are important for biological research.
“Our goal at DeepMind has always been to build AI and then use it as a tool to help accelerate the pace of scientific discovery itself, thereby advancing our understanding of the world around us,” said DeepMind Founder and CEO Demis Hassabis, PhD. “We used AlphaFold to generate the most complete and accurate picture of the human proteome. We believe this represents the most significant contribution AI has made to advancing scientific knowledge to date, and is a great illustration of the sorts of benefits AI can bring to society.”
AlphaFold is already helping scientists to accelerate discovery
The ability to predict a protein’s shape computationally from its amino acid sequence – rather than determining it experimentally through years of painstaking, laborious and often costly techniques – is already helping scientists to achieve in months what previously took years.
“The AlphaFold database is a perfect example of the virtuous circle of open science,” said EMBL Director General Edith Heard. “AlphaFold was trained using data from public resources built by the scientific community so it makes sense for its predictions to be public. Sharing AlphaFold predictions openly and freely will empower researchers everywhere to gain new insights and drive discovery. I believe that AlphaFold is truly a revolution for the life sciences, just as genomics was several decades ago and I am very proud that EMBL has been able to help DeepMind in enabling open access to this remarkable resource.”
AlphaFold is already being used by partners such as the Drugs for Neglected Diseases Initiative (DNDi), which has advanced their research into life-saving cures for diseases that disproportionately affect the poorer parts of the world, and the Centre for Enzyme Innovation (CEI) is using AlphaFold to help engineer faster enzymes for recycling some of our most polluting single-use plastics. For those scientists who rely on experimental protein structure determination, AlphaFold’s predictions have helped accelerate their research. For example, a team at the University of Colorado Boulder is finding promise in using AlphaFold predictions to study antibiotic resistance, while a group at the University of California San Francisco has used them to increase their understanding of SARS-CoV-2 biology.
The AlphaFold Protein Structure Database
The AlphaFold Protein Structure Database builds on many contributions from the international scientific community, as well as AlphaFold’s sophisticated algorithmic innovations and EMBL-EBI’s decades of experience in sharing the world’s biological data. DeepMind and EMBL’s European Bioinformatics Institute (EMBL-EBI) are providing access to AlphaFold’s predictions so that others can use the system as a tool to enable and accelerate research and open up completely new avenues of scientific discovery.
“This will be one of the most important datasets since the mapping of the Human Genome,” said EMBL Deputy Director General, and EMBL-EBI Director Ewan Birney. “Making AlphaFold predictions accessible to the international scientific community opens up so many new research avenues, from neglected diseases to new enzymes for biotechnology and everything in between. This is a great new scientific tool, which complements existing technologies, and will allow us to push the boundaries of our understanding of the world.”
In addition to the human proteome, the database launches with ~350,000 structures including 20 biologically-significant organisms such as E.coli, fruit fly, mouse, zebrafish, malaria parasite and tuberculosis bacteria. Research into these organisms has been the subject of countless research papers and numerous major breakthroughs. These structures will enable researchers across a huge variety of fields – from neuroscience to medicine – to accelerate their work.
The future of AlphaFold
The database and system will be periodically updated as we continue to invest in future improvements to AlphaFold, and over the coming months we plan to vastly expand the coverage to almost every sequenced protein known to science – over 100 million structures covering most of the UniProt reference database.
To learn more, please see the Nature papers describing our full method and the human proteome, and read the Authors’ Notes. See the open-source code to AlphaFold if you want to view the workings of the system, and Colab notebook to run individual sequences. To explore the structures, visit EMBL-EBI’s searchable database that is open and free to all.
Feedback from the scientific community
Jacques Dubochet, Nobel Laureate for Chemistry 2017, former Group Leader at EMBL
“I love to know that the collaboration between DeepMind and EMBL will make all the knowledge about protein structure open to all.”
Paul Nurse, Nobel Laureate for Physiology or Medicine 2001, Director of the Francis Crick Institute and Chair of EMBL Science Advisory Committee
“With this resource freely and openly available, the scientific community will be able to draw on collective knowledge to accelerate discovery, ushering in a new era for AI-enabled biology.”
Venki Ramakrishnan, Nobel Laureate for Chemistry 2009 and former President of the Royal Society
“This computational work represents a stunning advance on the protein-folding problem, a 50-year-old grand challenge in biology.”
Prof. Dame Janet Thornton, Director Emeritus of EMBL-EBI
“This contributes to our knowledge and understanding of living systems, with all the opportunities for humanity this will unlock.”
This post was originally published on EMBL-EBI News
DeepMind und EMBL veröffentlichen vollständige Datenbank mit vorausberechneten 3D Darstellungen aller menschlichen Proteine
DeepMind gab heute seine Partnerschaft mit dem Europäischen Laboratorium für Molekularbiologie (EMBL), Europas Vorzeigelabor für Biowissenschaften, bekannt, um die bisher vollständigste und genaueste Datenbank der rund 20 000 Proteine und ihrer vorausberechneten Strukturen aus dem menschlichen Genom, also das vollständige menschlichen Proteom, der wissenschaftliche Gemeinschaft frei zur Verfügung zu stellen. Die Datenbank und die Anwendung künstlicher/artifizieller Intelligenz (AI) bieten Strukturbiologen leistungsstarke neue Werkzeuge für die Untersuchung dreidimensionaler Strukturen von Proteinen. Sie eröffnen damit eine Fundgrube an Daten, die künftige Forschung und Erkenntnisse ermöglichen und eine neue Ära der AI-gestützten Biologie einläuten können.
Die Anerkennung von AlphaFold als Lösung für die 50 Jahre alte große Herausforderung der Proteinstrukturvorhersage im Dezember 2020 durch die Organisatoren des CASP-Benchmarks (Critical Assessment of Protein Structure Prediction) stellte einen signifikanter Durchbruch in der Forschung dar. Die AlphaFold-Proteinstrukturdatenbank baut auf dieser Innovation und den Entdeckungen von Generationen von WissenschaftlerInnen auf: von den frühen Pionieren der Proteinforschung und kristallographie bis hin zu den tausenden VorhersagespezialistInnen und StrukturbiologInnen, die seit Jahren Proteinstrukturen erforschen. Durch die Datenbank wird die Anzahl der hochpräzise dargestellten menschlichen Proteinstrukturen, die Forschenden zur Verfügung stehen, mehr als verdoppelt. AlphaFold erweitert damit das gesammelte Wissen über Proteinstrukturen drastisch. Ein besseres Verständnis dieser Bausteine des Lebens, die jedem biologischen Prozess in jedem Lebewesen zugrunde liegen, wird es Forschenden in den verschiedensten Bereichen ermöglichen, ihre Arbeit bedeutend zu beschleunigen.
Letzte Woche wurde die Methodik hinter der neuesten, hochinnovativen Version von AlphaFold, dem hochentwickelten AI-System, das Strukturvorhersagen ermöglicht, und dessen Open-Source-Code in der wissenschaftliche Fachzeitschrift Nature veröffentlicht. Die heutige Ankündigung fällt mit einer zweiten Veröffentlichung in der Nature zusammen, die nicht nur das umfassendste Bild der Proteine im menschlichen Proteom beschreibt, sondern auch die Proteinstrukturen 20 weiterer Organismen, die für die biologische Forschung bedeutend sind.
“Unser Ziel bei DeepMind war es immer, AI zu entwickeln und sie dann als Werkzeug zu nutzen, um das Tempo wissenschaftlicher Entdeckungen zu beschleunigen und damit unser Verständnis der Welt um uns herum zu verbessern”, sagte DeepMind-Gründer und CEO Demis Hassabis, PhD. “Wir haben AlphaFold verwendet, um das vollständigste und genaueste Bild des menschlichen Proteoms zu erstellen. Wir glauben, dass dies der bedeutendste Beitrag ist, den AI bisher zum Fortschritt wissenschaftlicher Erkenntnisse geleistet hat, und es ist ein großartiges Beispiel für die Art von Nutzen, den AI der Gesellschaft bringen kann.”
AlphaFold hilft WissenschaftlerInnen schon jetzt dabei, Entdeckungen zu beschleunigen
Die Möglichkeit, die Form eines Proteins nun rechnerisch auf Grund seiner Aminosäuresequenz vorherzusagen – anstatt sie experimentell durch mühsame und oft kostspielige Techniken zu bestimmen – hilft WissenschaftlerInnen bereits jetzt, in wenigen Monaten zu erreichen, was früher Jahre dauerte.
“Die AlphaFold-Datenbank ist ein perfektes Beispiel für den kontinuierlichen Erfolgszyklus von offener Wissenschaft”, sagte EMBL-Generaldirektorin Edith Heard. “AlphaFold wurde mit Datensätzen aus öffentlichen Ressourcen gefüttert, die von der wissenschaftlichen Gemeinschaft bereitgestellt wurden. Es ist also nur konsequent, dass seine Vorhersagen wiederum frei zugänglich gemacht werden. Die offene und freie Weitergabe der AlphaFold-Vorhersagen wird es Forschenden überall auf der Welt ermöglichen, neue Erkenntnisse zu gewinnen und Entdeckungen voranzutreiben. Ich gehe davon aus, dass AlphaFold eine wirkliche Revolution für die Biowissenschaften ist, so wie es die Genomik vor einigen Jahrzehnten war, und ich bin sehr stolz darauf, dass EMBL DeepMind dabei helfen konnte, den offenen Zugang zu dieser bemerkenswerten Ressource zu ermöglichen.”
AlphaFold wird bereits von Partnern wie der Drugs for Neglected Diseases Initiative (DNDi) eingesetzt, die damit ihre Forschung nach lebensrettenden Heilmitteln für Krankheiten vorantreibt, welche unverhältnismäßig stark wirtschaftlich benachteiligte Teilen der Welt betreffen. Das Centre for Enzyme Innovation (CEI) wiederum nutzt AlphaFold, um schnellere Enzyme für das Recycling einiger der umweltschädlichsten Einwegkunststoffe zu entwickeln. Auch WissenschaftlerInnen, die ihre Forschung auf die experimentelle Bestimmung von Proteinstrukturen basieren, helfen die Vorhersagen von AlphaFold bereits dabei, ihre Arbeit zu beschleunigen. So nutzt zum Beispiel ein Forschungsteam an der University of Colorado Boulder die AlphaFold-Vorhersagen, um Antibiotikaresistenzen zu untersuchen, während eine Gruppe an der University of California San Francisco sie verwendet, um ihr Verständnis der Biologie von SARS-CoV-2 zu verbessern.
Die AlphaFold-Proteinstruktur-Datenbank
Die AlphaFold Protein Structure Database baut sowohl auf vielen früheren Beiträgen der internationalen wissenschaftlichen Gemeinschaft auf, als auch auf den hochentwickelten algorithmischen Innovationen von AlphaFold und der jahrzehntelangen Erfahrung des European Bioinformatics Institute (EMBL-EBI) bei der freien Bereitstellung von weltweit gesammelten biologischen Daten. DeepMind und EMBL-EBI stellen den Zugang zu den Vorhersagen von AlphaFold offen zur Verfügung, damit die wissenschaftliche Gemeinschaft das System als Werkzeug dazu nutzen kann, weitere Forschung zu ermöglichen, zu beschleunigen und völlig neue Wege der wissenschaftlichen Entdeckung zu eröffnen.
“AlphaFold wird sich als einer der wichtigsten Datensätze seit der Kartierung des menschlichen Genoms herausstellen”, sagte der stellvertretende EMBL-Generaldirektor und EMBL-EBI-Direktor Ewan Birney. “Die AlphaFold-Vorhersagen der internationalen wissenschaftlichen Gemeinschaft offen zugänglich zu machen, eröffnet so viele neue Forschungsmöglichkeiten. Von vernachlässigten Krankheiten bis hin zu neuen Enzymen für die Biotechnologie und allem, was dazwischen liegt. Dies ist ein großartiges neues wissenschaftliches Werkzeug, das bestehende Technologien ergänzt und es uns ermöglichen wird, die Grenzen unseres Verständnisses der Welt zu erweitern.”
Zusätzlich zum menschlichen Proteom geht die Datenbank mit rund 350.000 weiteren Proteinstrukturen online, darunter Proteine von 20 biologisch bedeutsame Organismen wie der Fruchtfliege, der Maus, des Zebrafisches, des Malariaparasiten sowie von E.coli- und Tuberkulosebakterien. Die Forschung an diesen Organismen war bereits Grundlage zahlreicher bedeutender wissenschaftlicher Durchbrüche und entsprechender Veröffentlichungen. Ihre nun veröffentlichte Proteinstrukturen werden es Forschern in den verschiedensten Bereichen – von den Neurowissenschaften bis zur Medizin – ermöglichen, ihre Forschung schneller voranzutreiben.
Die Zukunft von AlphaFold
Die beteiligten Partner werden in den kommenden Monaten kontinuierlich in die Verbesserung von AlphaFold investieren und die Datenbank und das dahinterliegende System regelmäßig aktualisieren. So soll die Abdeckung auf fast alle sequenzierten Proteine, die der Wissenschaft bekannt sind, erweitert werden – über 100 Millionen Strukturen, die den Großteil der UniProt-Referenzdatenbank abdecken.
Weitere Informationen können Sie dem Fachartikel zum Thema in der Nature entnehmen, der die vollständige Methodik und das menschliche Proteom beschreibt sowie den Anmerkungen der Autoren. Besuchen Sie den Open-Source-Code zu AlphaFold, wenn Sie sich die Funktionsweise des Systems ansehen möchten und Colab notebook, um einzelne Sequenzen zu untersuchen. Um die Strukturen zu erforschen, besuchen Sie die Datenbank von EMBL-EBI, die offen und kostenfrei zugänglich ist.
